Hintergrund und Herstellung neuer Maismutanten
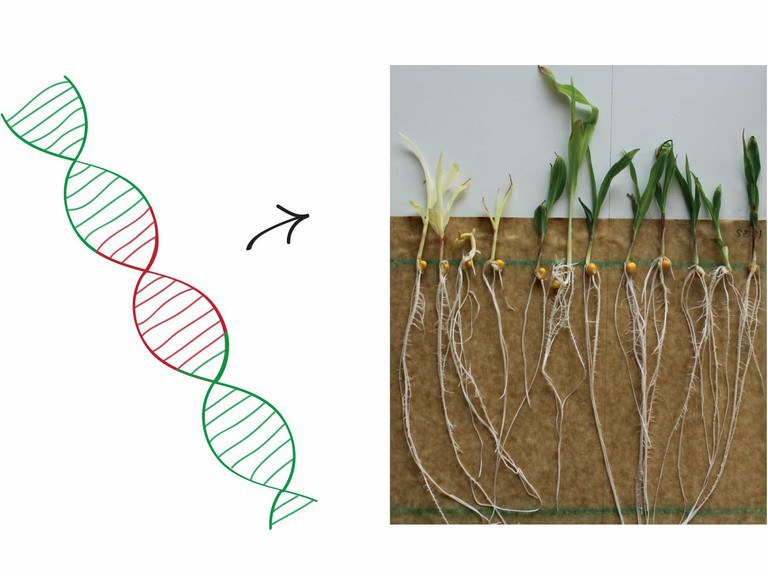
Bei der am Institut für Nutzpflanzenwissenschaft und Ressourcenschutz der Universität Bonn hergestellten Maismutanten-Kollektion handelt es sich um eine Bibliothek induzierter Mutationen in Mais (Zea mays), die mittels Insertionsmutagenese erzeugt wurde. Die Insertionsmutagenese ist eine Methode, bei der Mutationen auf natürliche Weise durch zufällige Integration von mobilen DNA-Fragmenten innerhalb eines Genoms erzeugt werden. Die durch mobile DNA-Fragmente getroffenen Gene werden häufig in ihrer Funktion ausgeschaltet. Resultierende Mutationen können dann erhebliche Auswirkungen z.B. auf das Wachstum, die Photosynthese (Abbildung 1) oder die Fruchtbarkeit der Maispflanzen haben.
Für die Erzeugung von Maismutanten nutzten Dr. Caroline Marcon und Kolleg*innen, aus der Arbeitsgruppe Crop Functional Genomics, Mutator (Mu)-Transposons, die auch als springende Gene bezeichnet werden. Zunächst wurden auf dem Campus Klein-Altendorf und in Endenich aktive Mu-Linien in verschiedene amerikanische (B73 und Co125) und europäische Mais-Inzuchtlinien (z.B. F7 und EP1) gekreuzt. „Diese Kreuzung war notwendig, da nur die Mu-aktive Mais-Linie das Enzym Transposase exprimiert, das die in den einzelnen Inzuchtlinien stillgelegten Mu-Transposons mobilisiert und dadurch Gene ausgeschaltet werden können“, sagt Dr. Caroline Marcon, die die Studie federführend geleitet hat. Im laufenden Projekt wurden bislang über 6.300 Mutator-induzierte Maisfamilien hergestellt, die nun sukzessive mittels Hochdurchsatzsequenzierung analysiert werden, um die neuen Mu-Insertionsstellen – und damit Mutationen – zu identifizieren. Maßgeblich beteiligt an der Identifizierung neuer Insertionsstellen sind Kooperationspartner aus der Arbeitsgruppe Crop Bioinformatics, die von Prof. Dr. Heiko Schoof geleitet wird.
Für die jetzt veröffentlichte Studie wurden 1.152 transposon-induzierte Maisfamilien im genetischen Hintergrund der Inzuchtlinie B73 sequenziert. Dabei konnten etwa 41.000 vererbbare Mu-Insertionen nachgewiesen werden, die mehr als 16.300 (37%) der derzeit annotierten Maisgene betreffen. In der Studie verglichen Caroline Marcon und ihre Kolleg*innen erstmalig sämtliche Mu-inserierten Gene von BonnMu mit solchen der amerikanischen UniformMu Ressource. Diese UniformMu Kollektion wurde mit Hilfe einer anderen Mais-Inzuchtlinie (W22) hergestellt. Bei dem Vergleich fanden die Wissenschaftler*innen heraus, dass (1) Insertions-Hotspots zwischen den beiden Mais-Genotypen B73 und W22 konserviert sind und, dass (2) die Mehrzahl (75%) der Mu-Insertionsstellen näher am Transkriptionsstartpunkt als am Startcodon der betroffenen Gene liegt. Letzteres war insofern interessant, als dass aktive Gene im Bereich des Transkriptions-startpunkts häufig durch offenes Chromatin gekennzeichnet sind. Als Chromatin wird der Komplex aus DNA und Protein bezeichnet, aus dem die Chromosomen bestehen. Aufgelockertes Chromatin könnte nicht nur leicht für die Transkriptionsmaschinerie, sondern auch leicht für Mu-Transposons zugänglich sein. Diese These wird nun durch die in der Studie identifizierte hohe Anzahl von Mu-Insertionsstellen am Transkriptionsstart unterstützt.
Für fast 50% aller Maisgene gibt es inzwischen BonnMu-Insertionsmutationen
Durch fortlaufende Sequenzierungen der mutagenisierten Maisfamilien im BonnMu-Projekt konnten inzwischen über 95.000 Insertionsstellen identifiziert werden, die 21.613 (49%) der 44.117 Maisgene betreffen. „Das Ziel des Projekts ist es aber,“ sagt Projektleiterin Caroline Marcon, „möglichst alle Gene des Maisgenoms durch Mu-Insertionen auszuschalten. Phänotypen der Maismutanten können dann helfen, die Funktion von Genen aufzuklären. Denn bislang sind nur einige 100 Maisgene funktionell beschrieben.“
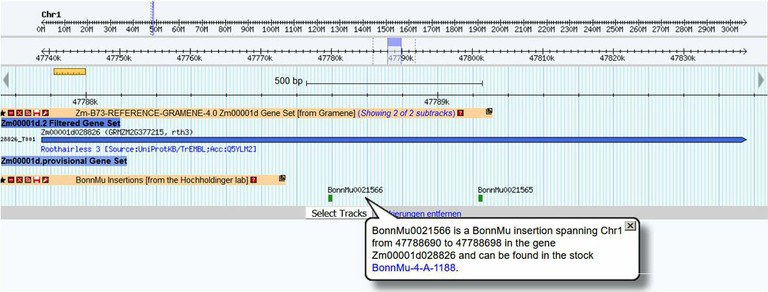
Die fortlaufende Zusammenarbeit mit Wissenschaftler*innen der USA ermöglicht die regelmäßige Einpflegung der BonnMu-Insertionsstellen in die Online-Datenbank „MaizeGDB“ (maizegdb.org), eine der wichtigsten Datenbanken für Maisforscher*innen. Im Webbrowser von „MaizeGDB“ können die BonnMu-Insertionsstellen in den einzelnen Genen visualisiert werden (Abbildung 2). Außerdem sind dort auch Keimlings-Fotos der mutagenisierten Maisfamilien hinterlegt (Abbildung 1). „Interessierte Wissenschaftler*innen können auf die öffentliche Datenbank zugreifen und dort gezielt nach Mu-Insertionen suchen, die auf ihrer Forschungsagende stehen. Und auf Anfrage stellen wir die entsprechenden Maismutanten für funktionelle Gen-Analysen zur Verfügung,“ sagt Caroline Marcon abschließend.
Beteiligte Institutionen und Förderung
An diesem Projekt waren internationale Wissenschaftler*innen der Universitäten Florida und Missouri, sowie das Landwirtschaftsministerium der Vereinigten Staaten beteiligt. Die deutsche Forschungsgemeinschaft DFG unterstützte das Projekt finanziell.
Publikation
Marcon C, Altrogge L, et al., (2020) BonnMu: a sequence-indexed resource of transposon induced maize mutations for functional genomics studies, Plant Physiology, DOI: https://doi.org/10.1104/pp.20.00478.